Identificación de la región pseudoautosómica del cromosoma X en ovinos y pre-dicción del sexo mediante el ensamblado ARS-UI_Ramb_v2.0
DOI:
https://doi.org/10.31285/AGRO.29.1587Palabras clave:
ARS-I_Ramb_v2.0, región pseudoautosómica, predicción del sexo, cromosoma XResumen
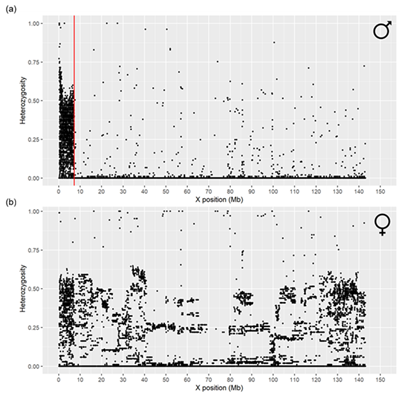
La determinación de sexo mediante genotipado es eficaz para verificar el sexo declarado en el pedigrí. Como los machos poseen un solo cromosoma X, no se deberían observar genotipos heterocigotos en la región no pseudoautosómica (nPAR). Por lo tanto, es fundamental establecer claramente los límites de la región pseudoautosómica (PAB) para una correcta identificación del sexo. Aunque se recomienda utilizar SNP de los cromosomas X e Y, el chip de genotipado utilizado en este estudio no contenía marcadores del cromosoma Y. El objetivo de este trabajo fue identificar la región pseudoautosómica (PAR) en el cromosoma X en ovinos utilizando el ensamblado ARS-UI_Ramb_v2.0 y evaluar un método para la predicción de sexo basado únicamente en los SNP del cromosoma X. Para definir la región PAR, se utilizó una población de 210 ovinos, genotipados con el Ovine Infinium® HD SNP BeadChip, y para la validación, 229 ovinos, genotipados con el OvineSNP50 BeadChip. Después del control de calidad, se observó que la región PAR se extendía desde 0 a 7,24 Mb, identificada mediante SNP que exhibieron altas tasas de heterocigosidad en machos. El enfoque propuesto, que utiliza únicamente los SNP del cromosoma X, alcanzó una exactitud de 94% en la predicción de sexo, con una precisión de 99% en machos y de 89% en hembras. Este estudio demostró que es posible predecir el sexo en ovinos utilizando únicamente los SNP de la región nPAR del cromosoma X. Este es el primer trabajo en aplicar este método utilizando el ensamblado ARS-UI_Ramb_v2.0.
Palabras clave: ARS-I_Ramb_v2.0, región pseudoautosómica, predicción del sexo, cromosoma X
Identificação da região pseudoautossômica do cromossomo X em ovinos e predição de sexo usando a montagem do genoma ARS-UI_Ramb_v2.0
Resumo
A determinação do sexo por genotipagem é uma ferramenta eficiente para verificar o sexo registrado. Como os machos possuem apenas um cromossomo X, os genótipos heterozigotos não devem estar presentes na região não pseudoautossômica (nPAR) do cromossomo X. Portanto, determinar os limites da região pseudoautossômica (PAB) é necessário para uma predição confiável do sexo. Embora seja recomendado usar SNP dos cromossomos X e Y, o chip SNP que usamos não continha marcadores do cromossomo Y. Este estudo teve como objetivo determinar a região pseudoautossômica (PAR) no cromossomo X em ovinos usando o genoma ARS-UI_Ramb_v2.0 e testar um método para predição de sexo baseado apenas em SNP do cromossomo X. Para definição da região PAR foi utilizada uma população de treinamento de 210 ovinos genotipados com o Ovine Infinium® HD SNP BeadChip e para validação, 229 ovinos genotipados com o OvineSNP50 BeadChip. Após controle de qualidade, observou-se que a região PARse estendia de 0 a 7,24 Mb, identificada por SNP que exibiam altas taxas de heterozigosidade em machos. A abordagem proposta, que usa apenas SNPs do cromossomo X, atingiu uma exatidão de predição de sexo de 94%, com taxas de precisão de 99% para machos e 89% para fêmeas. Este estudo mostrou que a predição de sexo em ovinos pode ser feita usando apenas SNP da região nPAR do cromossomo X. Este é o primeiro estudo que usou esta abordagem especificamente usando o genoma ARS-UI_Ramb_v2.0.
Descargas
Referencias bibliográficas
Aguilar I, Misztal I, Tsuruta S, Legarra A, Wang H. PREGSF90 – POSTGSF90: computational tools for the implementation of single-step genomic selection and genome-wide association with ungenotyped individuals in BLUPF90 programs. In: Proceedings 10th World Congress of Genetics Applied to Livestock Production. Vancouver: American Society of Animal Science; 2014. p. 680.
Baylis MS, Wayte DM, Owen JB. An XO/XX mosaic sheep with associated gonadal dysgenesis. Res Vet Sci. 1984;36(1):125-6. DOI: https://doi.org/10.1016/S0034-5288(18)32013-7
Berkowic Y, Edwards LA, Busch R, Pérez-Nogués M, Stevens SE, Booth M, McNabb BR. Male pseudohermaphrodite in a Shetland cross sheep: a case report. Clinical Theriogenology. 2017;9(4):605-13.
Berry DP, Spangler ML. Animal board invited review: practical applications of genomic information in livestock. Animal. 2023;17(11):100996. Doi: 10.1016/j.animal.2023.100996. DOI: https://doi.org/10.1016/j.animal.2023.100996
Bilton TP, Chappell AJ, Clarke SM, Brauning R, Dodds KG, McEwan JC, Rowe SJ. Using genotyping-by-sequencing to predict gender in animals. Anim Genet. 2019;50(3):307-10. Doi: 10.1111/age.12782. DOI: https://doi.org/10.1111/age.12782
Bruere AN, Marshall RB, Ward DP. Testicular hypoplasia and XXY sex chromosome complement in two rams: the ovine counterpart of Klinefelter's syndrome in man. J Reprod Fertil. 1969;19(1):103-8. Doi: 10.1530/jrf.0.0190103. DOI: https://doi.org/10.1530/jrf.0.0190103
Champely S. pwr: basic functions for power analysis [Internet]. Version 1.3-0. 2020 [cited 2025 May 27]. Available from: https://CRAN.R-project.org/package=pwr
Ciappesoni G, Marques C, Navajas EA, Peraza P, Carracelas B, Vera B, Van Lier E, De Barbieri I, Salada S, Monzalvo C, Castells D. Breeding for sheep parasite resistance in extensive production systems in Uruguay: from phenotype to genotype. In: Viljoen G, Garcia Podesta M, Boettcher P, editors. International Symposium on Sustainable Animal Production and Health - Current status and way forward. Rome: FAO; 2023. p. 224-36.
Johnson T, Keehan M, Harland C, Lopdell T, Spelman RJ, Davis SR, Rosen BD, Smith TPL, Couldrey C. Identification of the pseudoautosomal region in the Hereford bovine reference genome assembly ARS-UCD1.2. J Dairy Sci. 2019;102(4):3254-8. Doi: 10.3168/jds.2018-15638. DOI: https://doi.org/10.3168/jds.2018-15638
Katoh K, Rozewicki J, Yamada KD. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization. Brief Bioinform. 2019;20(4):1160-6. Doi: 10.1093/bib/bbx108. DOI: https://doi.org/10.1093/bib/bbx108
Kuhn M. Building Predictive Models in R Using the caret Package. J Stat Softw. 2008;28(5):1-26. Doi: 10.18637/jss.v028.i05 DOI: https://doi.org/10.18637/jss.v028.i05
Liu R, Low WY, Tearle R, Koren S, Ghurye J, Rhie A, Phillippy AM, Rosen BD, Bickhart DM, Smith TPL, Hiendleder S, Williams JL. New insights into mammalian sex chromosome structure and evolution using high-quality sequences from bovine X and Y chromosomes. BMC Genomics. 2019;20(1):1000. Doi: 10.1186/s12864-019-6364-z. DOI: https://doi.org/10.1186/s12864-019-6364-z
McClure MC, McCarthy J, Flynn P, McClure JC, Dair E, O'Connell DK, Kearney JF. SNP data quality control in a national beef and dairy cattle system and highly accurate SNP based parentage verification and identification. Front Genet. 2018;9:84. Doi: 10.3389/fgene.2018.00084. DOI: https://doi.org/10.3389/fgene.2018.00084
Medrano JF, Aasen E, Sharrow L. DNA extraction from nucleated red blood cells. Biotechniques. 1990;8(1):43.
Olagunju TA, Rosen BD, Neibergs HL, Becker GM, Davenport KM, Elsik CG, Hadfield TS, Koren S, Kuhn KL, Rhie A, Shira KA, Skibiel AL, Stegemiller MR, Thorne JW, Villamediana P, Cockett NE, Murdoch BM, Smith TPL. Telomere-to-telomere assemblies of cattle and sheep Y-chromosomes uncover divergent structure and gene content. Nat Commun. 2024;15(1):8277. Doi: 10.1038/s41467-024-52384-5. DOI: https://doi.org/10.1038/s41467-024-52384-5
Ramos Z, Garrick DJ, Blair HT, De Barbieri I, Ciappesoni G, Montossi F, Kenyon PR. Genetic trends for production and reproduction traits in ultrafine Merino sheep of Uruguay. J Anim Breed Genet. Forhcoming 2025. Doi: 10.1111/jbg.12937. DOI: https://doi.org/10.1111/jbg.12937
Raudsepp T, Chowdhary BP. Chromosome aberrations and fertility disorders in domestic animals. Annu Rev Anim Biosci. 2016;4:15-43. Doi: 10.1146/annurev-animal-021815-111239. DOI: https://doi.org/10.1146/annurev-animal-021815-111239
Raudsepp T, Das PJ, Avila F, Chowdhary BP. The pseudoautosomal region and sex chromosome aneuploidies in domestic species. Sex Dev. 2012;6(1-3):72-83. Doi: 10.1159/000330627. DOI: https://doi.org/10.1159/000330627
Ristanic M, Stanisic L, Maletic M, Glavinic U, Draskovic V, Aleksic N, Stanimirovic Z. Bovine foetal sex determination-Different DNA extraction and amplification approaches for efficient livestock production. Reprod Domest Anim. 2018;53(4):947-54. Doi: 10.1111/rda.13193. DOI: https://doi.org/10.1111/rda.13193
Ryan CA, Purfield DC, Matthews D, Canedo-Ribeiro C, Valldecabres A, Berry DP. Prevalence of sex-chromosome aneuploidy estimated using SNP genotype intensity information in a large population of juvenile dairy and beef cattle. J Anim Breed Genet. 2024;141(5):571-85. Doi: 10.1111/jbg.12866. DOI: https://doi.org/10.1111/jbg.12866
Shihabi M, Lukic B, Cubric-Curik V, Brajkovic V, Oršanić M, Ugarković D, Vostry L, Curik I. Identification of selection signals on the X-Chromosome in East Adriatic Sheep: a new complementary approach. Front Genet. 2022;13:887582. Doi: 10.3389/fgene.2022.887582. DOI: https://doi.org/10.3389/fgene.2022.887582
Wickham H. ggplot2: elegant graphics for data analysis [Internet]. [place unknown]: The tidyverse team; [cited 2025 May 27]. Available from: https://ggplot2.tidyverse.org
Zartman DL, Hinesley LL, Gnatkowski MW. A 53,X female sheep (Ovis aries). Cytogenet Cell Genet. 1981;30(1):54-8. Doi: 10.1159/000131589. DOI: https://doi.org/10.1159/000131589
Zhang I, Couldrey C, Sherlock R. Using genomic information to predict sex in dairy cattle. Proc N Z Soc Anim Prod. 2016;76:26-30.
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2025 Agrociencia Uruguay

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |